用“分子支架”设计人工酶
作者: aeks | 发布时间: 2025-12-04 12:05 | 更新时间: 2025-12-04 12:05
学科分类: 化学工程与技术 生物学 生物工程 计算机科学与技术
天然酶是化学家不可或缺的工具,在化学反应中具有无与伦比的精准度和选择性。然而,寻找具有所需活性的天然酶可能需要大量资源和筛选能力。近年来,计算蛋白质设计已能创造针对多种反应的生物催化剂,但当前酶设计方法常被忽视的局限性是所设计生物催化剂的初始催化速率较低。即使有最新进展,生成高效生物催化剂的现有模式仍是通过高通量筛选和定向进化来弥补初始效率的不足。尽管这种方法有价值且稳健,但不适用于快速创建自然界中不存在或难以通过高通量筛选获得的新型高效催化剂,因此需要提高“一步到位”设计高效酶的能力。
从头酶设计策略通常基于酶催化的过渡态模型,即活性位点的官能团通过稳定反应过渡态来加速化学反应。早期计算设计的酶虽能催化化学反应,但催化速率低,常需大量筛选,主要限制因素之一是催化侧链在活性位点的放置精度。随着结构预测和蛋白质设计工具的革新,研究者尝试解决这一问题。
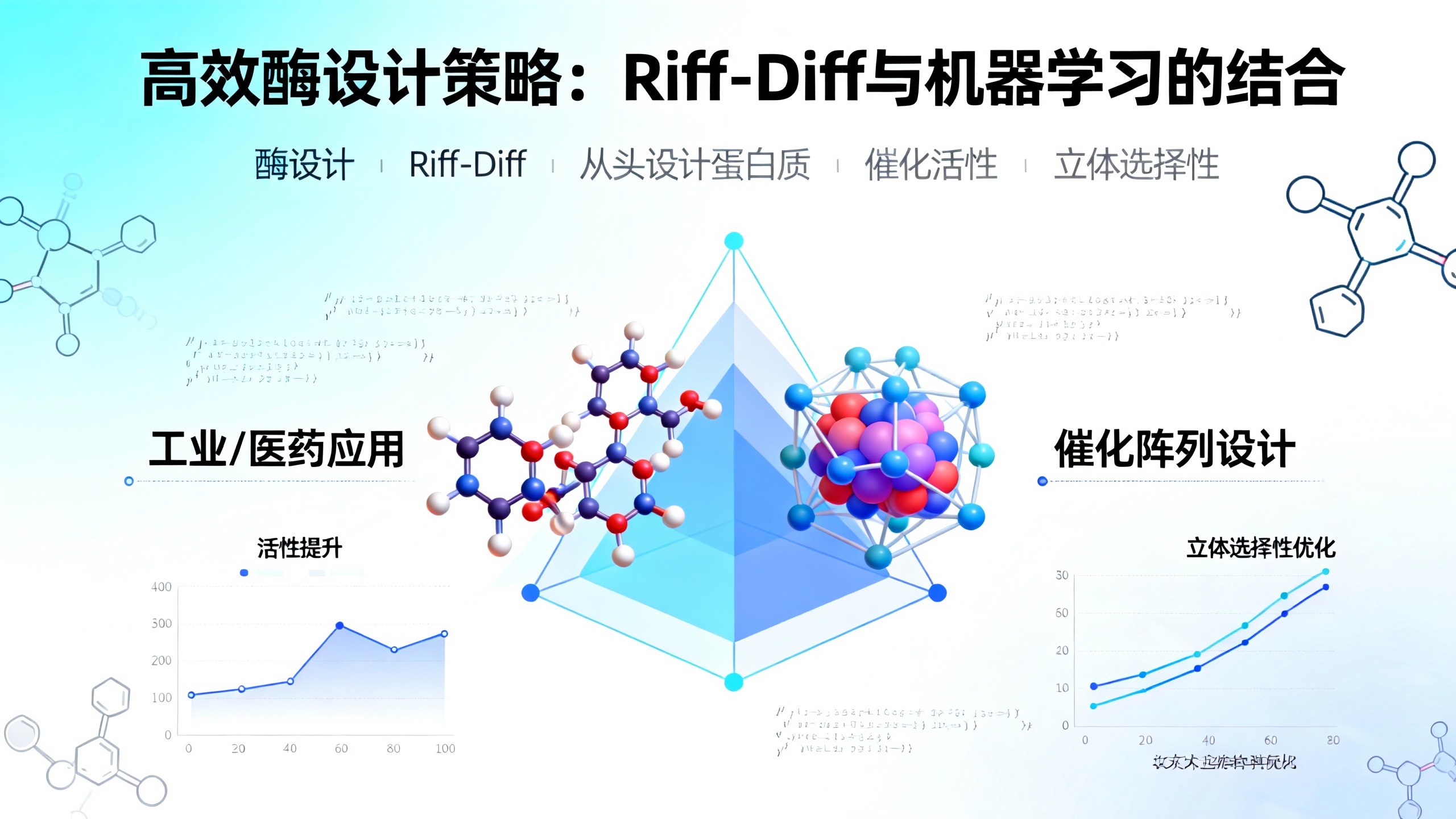
本研究旨在通过在从头蛋白质骨架中整合三个不同的催化中心,“一步到位”创建高效生物催化剂,为此开发了Riff-Diff方法。该方法专注于主链-侧链片段的精确放置、催化阵列的设计精度以及定制结合口袋的设计。为测试其有效性,研究者基于先前报道的高度优化催化四联体设计了逆羟醛反应酶,并基于两个不同的进化活性位点阵列设计了Morita-Baylis-Hillman(MBH)反应酶。
Riff-Diff的核心包括:构建人工基序库(将催化阵列的氨基酸嵌入螺旋片段,通过旋转异构体反转生成物理上合理的基序)、确保结合口袋形成(添加α螺旋作为结合口袋入口通道占位符,使生成的口袋模拟天然酶特征)、主链优化(迭代优化扩散后的酶主链,结合LigandMPNN、FastRelax、ESMFold等工具)。
在逆羟醛反应中,Riff-Diff设计的35个酶中32个活性高于阴性对照,7个设计的产物生成速率远高于筛选平均值。其中RAD29和RAD35的催化效率接近经广泛进化的酶,且具有立体选择性(RAD35对(R)-底物的对映体过量值达99%)和高热稳定性(多数在90°C以上仍保持折叠)。晶体结构显示活性位点设计精度达近原子水平,但活性不仅取决于侧链位置,构象灵活性也很重要。
在MBH反应中,基于两个进化活性位点阵列设计的酶中,94%和93.3%的设计活性高于背景水平,MBH48的催化活性优于经8轮定向进化筛选的变体,且副产物少。晶体结构证实其主链和活性位点与设计模型高度一致。
结论:Riff-Diff能“一步到位”设计催化(逆)羟醛和MBH反应的酶,活性超过以往设计,可与多轮定向进化酶媲美,且筛选序列少。该方法为减少繁琐的高通量筛选迈出重要一步,但仍面临活性预测(如RMSD等结构相似性指标不能准确预测活性)和构象动力学控制等挑战。未来需整合动态建模工具,以实现对活性位点动态行为的精准调控。