利用DNA三向交叉结构构建复杂的多样化DNA序列
作者: aeks | 发布时间: 2026-01-22 09:01 | 更新时间: 2026-01-22 09:01
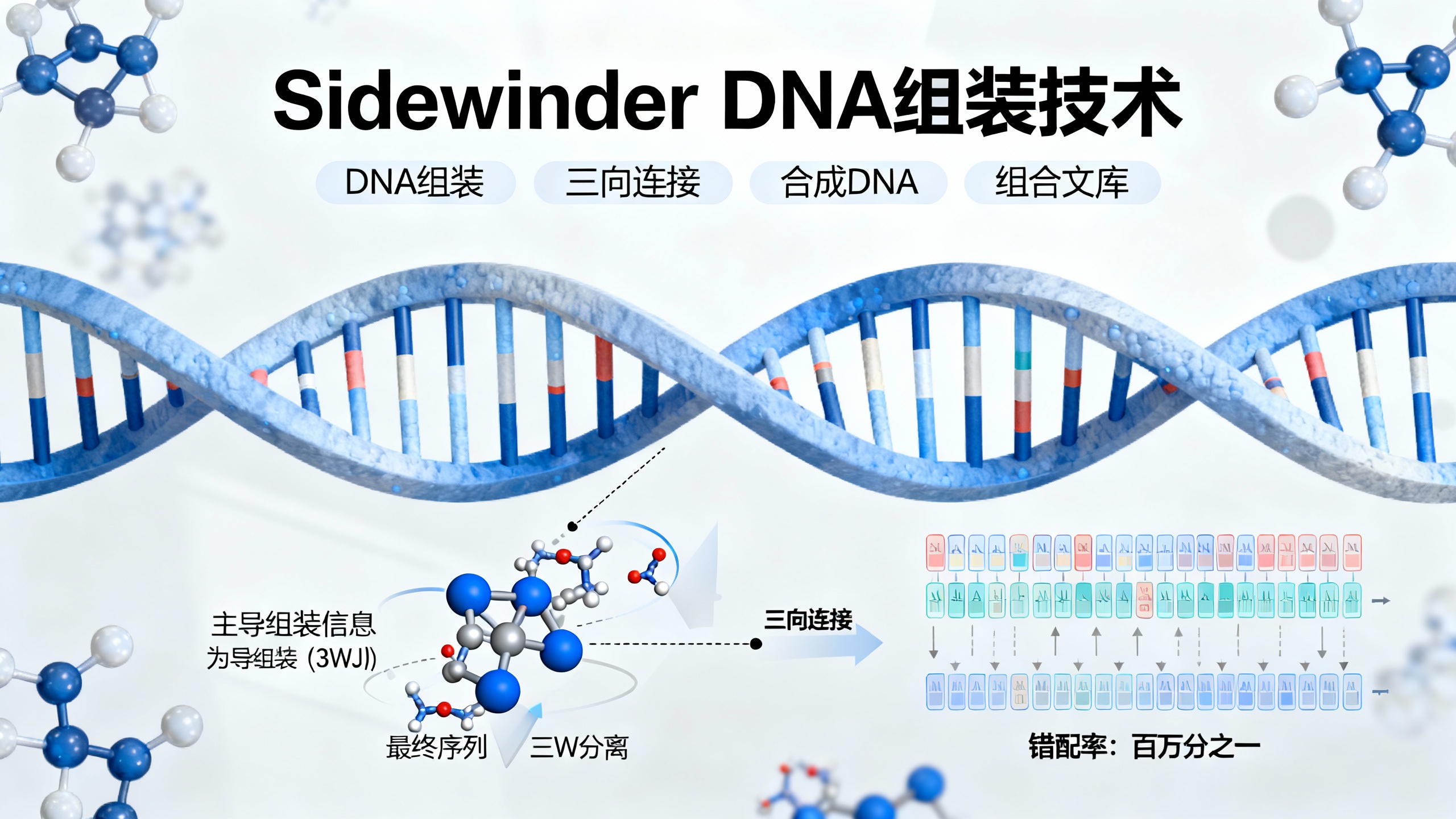
从头合成全新DNA序列对生物工程和研究至关重要,但目前长复杂DNA序列及文库的合成能力落后于测序和编辑技术。现有DNA组装技术均依赖最终构建体中的序列信息来指导DNA分子间的组装,这导致无法在不影响最终序列的前提下专门优化组装序列。为解决这一问题,研究人员开发了名为Sidewinder的新型DNA组装技术,它利用DNA三向连接(3WJ)将指导组装的信息与最终组装序列分离。
Sidewinder的核心创新在于依靠第三条独特的螺旋(Sidewinder螺旋)编码的信息,通过形成3WJ来指导DNA片段间的组装。该螺旋不参与最终组装序列,因此消除了对组装位置、序列类型和片段数量的限制。组装片段包含末端二级结构: toehold(t/t*,类似传统的双链突出端,存在于最终产物中)和Sidewinder条形码(b/b*,构成3WJ中的Sidewinder螺旋,不存在于最终产物中,且经过高度优化)。在高于toehold解链温度的条件下,长条形码的高解链温度指导片段特异性结合,形成3WJ后通过连接酶连接缺口,最后去除条形码螺旋,恢复传统双链连接,完成无痕组装。
实验证明,Sidewinder具有变革性:能稳健准确地组装40片段的多片段结构;成功构建高GC含量(如人类APOE基因,GC含量70%)和高重复序列(如蚕丝蛋白h-fibroin)的复杂DNA;可在同一反应中平行组装多个不同基因(如mScarlet、mGL和aeBlue);能构建在基因全长有大量多样化位点的组合文库(如eGFP基因的17个位点多样化,理论库容量442,368种变体,覆盖率超91.7%)。其3WJ处的错配率约为百万分之一,显著优于传统方法。
Sidewinder克服了传统技术的固有局限,为合成基因组、生物材料、蛋白质工程等领域提供了高效可靠的DNA组装工具,有望推动合成生物学及相关领域的发展。
标签: DNA组装 Sidewinder 三向连接 合成DNA 组合文库