候选基因调控元件的扩展名录
作者: aeks | 发布时间: 2026-01-09 12:01 | 更新时间: 2026-01-09 12:01
学科分类: 生物医学工程
哺乳动物基因组是DNA编码指令的庞大储存库,通过复杂调控机制控制细胞功能。其中核心的顺式调控元件(CREs)是一类非编码DNA序列,负责调控邻近基因的转录。它们通常与开放染色质及特定组蛋白修饰相关,包含转录因子等染色质相关蛋白的结合位点,这些蛋白相互作用并与转录机器协同调控基因表达。理解CREs的生物学背景和功能,对于解读基因组功能及其对人类健康与疾病的影响至关重要。
ENCODE项目通过系统识别和注释人类与小鼠基因组中的功能元件,为基因调控研究做出了重大贡献。该联盟在数万次高通量功能基因组学实验中,全面绘制了生化特征图谱,并以此注释CREs,包括ENCODE第三阶段(ENCODE3)的cCRE注册库。
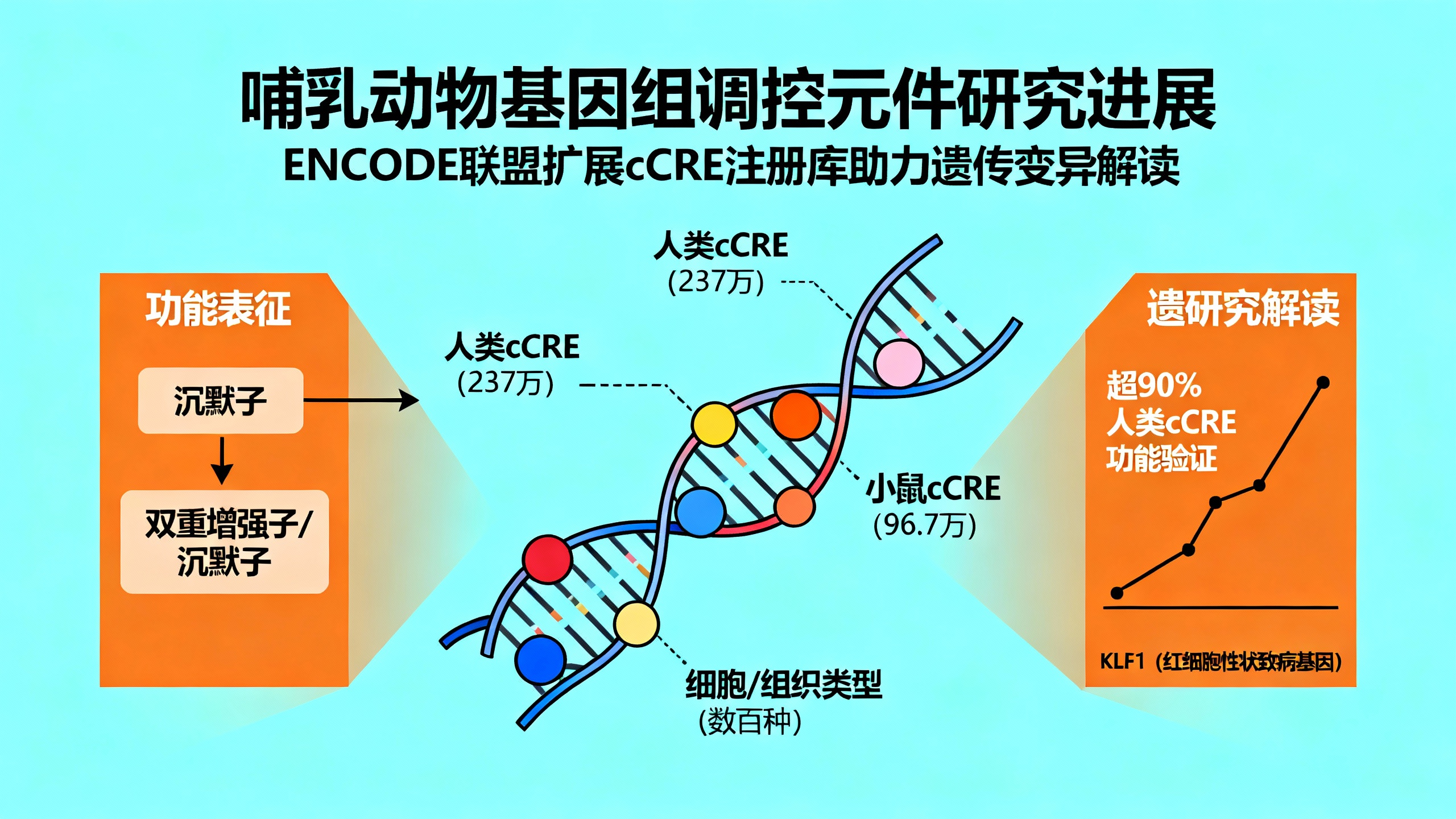
本文将cCRE注册库扩展至237万个人类和96.7万个小鼠元件。这一扩展得益于ENCODE第四阶段(ENCODE4)产生的新数据集和改进的计算方法,使其成为目前最广泛的CRE资源之一。更新后的注册库涵盖1679种独特的生物样本(包括组织、细胞类型和细胞状态),跨越42个人体器官和组织,增强了对广泛生物学背景下基因调控的理解。
除增加注释元件数量外,更新后的注册库还整合了超过97%人类cCRE的功能表征数据,揭示了序列特征如何影响调控活性,并发现了新的cCRE功能亚类。例如,研究人员识别出数千个沉默子cCRE,其中许多在不同细胞背景下可作为增强子发挥作用;还定义了MAFF和MAFK结合的cCRE作为动态增强子,在应激响应条件下可被激活。这些分析共同凸显了CRE功能的多样性和背景依赖性。
将扩展后的注册库与ENCODE百科全书的其他注释整合,能够系统解读遗传变异并识别性状相关基因。以红细胞性状为例,通过该注册库发现KLF1是一个新的致病基因,展示了其在遗传研究中的实用价值。
ENCODE4使用四种功能测定法(全基因组STARR-seq、大规模平行报告基因测定(MPRA)、CRISPR扰动测定和转基因小鼠增强子测定)测试了数百万基因组区域的活性。近97%的人类cCRE至少在一种细胞背景下接受了至少一种实验的测试,其中28%在至少一种实验中显示出显著活性。研究还开发了CAPRA方法,从RNA:DNA比率计算cCRE特异性STARR分数,解析增强子和沉默子活性。
在沉默子研究方面,团队以REST结合的神经元限制性沉默子元件(NRSEs)为起点,定义了REST+ cCREs,并将其分为两类:REST+增强子/沉默子(在神经元中为增强子,在非神经元中为沉默子)和REST+沉默子(仅在非神经元中起沉默作用)。利用STARR-seq数据还识别出STARR沉默子cCREs,它们富集抑制因子基序,邻近基因表达较低,参与在非表达背景下抑制组织特异性程序。
此外,MAFF和MAFK结合的TF cCREs被发现是一类潜在的刺激响应增强子,它们虽染色质可及性低,但在特定生物样本中显示高活性,邻近发育和信号转导相关基因。
该扩展的cCRE注册库为研究调控基因组及其对健康和疾病的影响提供了宝贵资源,支持从基因调控机制研究到疾病相关基因识别等多种科学探索。